近日,北京的大学深圳研究生院叶涛课题组通过应用生源合成规则成功预测了聚酮类天然产物eucalactam B的绝对立体构型,并通过全合成,利用Paterson Aldol以及CM等反应,以最长线性步骤19步,6.0%的收率完成了eucalactam B的首例全合成。此工作成功地验证了生源合成规则的有效性,对此类天然产物的结构鉴定和全合成有着重要的指导意义。
Eucalactam B为2020年由高锦明课题组从苦楝(Melia azedarach)内生真菌 Diaporthe eucalyptorum KY-9 培养提取物中分离得到的一种大环聚酮肽类化合物。其结构为一个拥有8个手性中心,由3个天然氨基酸(2个甘氨酸及1个L-苏氨酸)和一个不饱和脂肪羟基酸单元组成的21元大环酯肽。在分离和鉴定过程中,高锦明课题组通过半合成法以及Marfey法等化学手段,确认了其中苏氨酸的L构型以及C-13, C-15 -OH的syn相对构型。但是,由于分离产物的量有限以及分子柔性较大,无法通过XRD等方法确定其他手性中心的绝对构型。此外,Y. Marin-Felix等人在对Diaporthe africana培养物的分离鉴定工作中,同样发现了与eucalactam B类似的化合物。经过与前文高锦明课题组的分离产物进行谱图比对,证实二者为同一化合物。相似地,Y. Marin-Felix等人亦未能确定eucalactam B中其余手性中心的绝对立体构型。
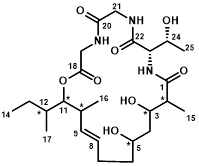
图1 最初报导的eucalactam B 的结构
来自真菌的高度还原聚酮合成酶(HR-PKSs)在聚酮类化合物的生源合成中起到了重要作用。这些酶通过催化延长线性脂肪酸链作为构建聚酮化合物的基本构建单元。在此基础上,HR-PKSs的结构特性可以精确控制聚酮链上的立体构型,从而得到特定构型的天然产物分子。2021年,叶涛课题组与北海道大学Oikawa课题组共同提出了此类聚酮化合物的生源合成规则。基于此规则,可以预测此类真菌源的聚酮化合物的绝对构型。如图2a所示,定义酰基侧为Rc,聚酮链初始延长侧为Rm,基团大小顺序为-OH>Rc>Rm>Me>H,此时从最小基团的对侧观察,基团从大到小呈顺时针排布则为oR构型,反之为oS构型。羰基还原酶将羰基还原为oR构型,烯酮还原酶将双键还原成oS构型,甲基转移酶则引入一个oR构型的甲基。
eucalactam B分子结构中有6个手性中心绝对构型未定。由于存在一对确定的相对构型,故其尚存在32种可能的结构。因此,通过单纯化学合成的手段揭示eucalactam B的真实立体结构存在着巨大的挑战。在这里,叶涛课题组在此借助上述提出的生源合成规律,可以推测到eucalactam B的结构如图2b所示:

图2. a) 对oR 和 oS立体构型的定义 b) 基于生源合成规则对eucalactam B 构型的预测。 图例:AT:酰基转移酶;MT:甲基转移酶;KR:羰基还原酶;ER:烯酮还原酶;KS:羰基合成酶;DH:脱水酶
在此基础上,作者通过借助全合成的手段来验证上述预测结构的准确性。
逆合成分析如图3所示。天然产物1可以通过二肽片段3和片段2通过接肽反应和大环内酰胺化反应得到。片段二又可拆分为聚酮链4和甘氨酸。4则可通过Paterson Aldol反应和CM反应拆分为已知片段5和6。
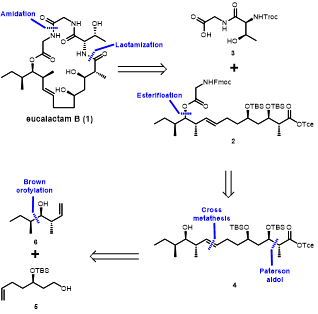
图3 天然产物eucalactam B的逆合成分析
该团队首先聚焦聚酮链右侧片段的合成。起始片段5可通过D-天冬氨酸由6步已知反应获得。经Dess-Martin氧化成醛后与已知化合物8发生Paterson Aldol反应得到单一异构体9。9经TBS保护得到化合物10后经一锅法还原-醇解、氧化裂解和Pinnick氧化后得到羧酸11,再经过Tce保护后得到右侧片段12。

图4 片段12的合成
随后化合物12可与已知片段6发生CM反应得到聚酮链4。随后,通过与Fmoc-甘氨酸发生Steglich酯化得到化合物2。2在DBU条件下与二肽3进行一锅法接肽得到关环前体13。13在锌粉和弱酸性条件下同时脱保护后在HATU催化下进行大环内酰胺化得到化合物14,再脱除硅保护基得到化合物1。经核磁、质谱、旋光等数据比对,化合物1的表征数据与天然产物eucalactam B 的数据高度吻合,证明了前文提及的生源合成规则在真菌源聚酮类天然产物结构预测中的正确性。

图5 天然产物eucalactam B (1)的合成
北京大学深圳研究生院博士研究生王辰齐为该论文的第一作者,该研究得到了国家自然科学基金和深圳市高校可持续发展支持基金的支持。
原文链接:https://pubs.rsc.org/en/content/articlelanding/2024/qo/d4qo01777c